Manipuler des données volumineuses avec Arrow & DuckDB
Problèmes de mémoire pour analyser vos données ?
On travaille de plus en plus avec des jeux de données de grande taille, et nécessitant une interopérabilité entre plateformes.
On doit alors gérer un casse-tête, entre le format de stockage, d’échange, et l’analyse de données qui ne tiennent pas en mémoire.
En effet :
- Comment analyser 100 Gb de données sur une PC qui n’a que 16 Gb de RAM ?
- Quel format utiliser pour des données qui peuvent être analysées par des équipes variées utilisant R, Python ou Julia ?
- Ou des application développés en C++, Java ou Rust?
- Indépendamment de la mémoire, comment réduire l’espace utilisé sur le disque ?
- Pourquoi charger toutes les données si je souhaite faire une analyse que d’une sous-catégorie ?
On utilise généralement le format .csv qui est facile à lire, même par un humain, et peut être utilisé dans tout langage et sur tous les systèmes. Cependant, ce format n’est pas compressé, et prend donc de la place sur disque. Il va nécessiter d’être chargé intégralement en mémoire même si on souhaite faire une analyse sur un sous ensemble des données. Cela a un coût aussi bien en stockage qu’en temps d’analyse.
Apache Arrow et DuckDB permettent de répondre à ces problèmes, sont capable d’interagir entre eux, et disposent de librairies pour la plupart des langages. Ce sont deux outils qui vont devenir indispensables à connaître pour la collecte, le stockage de données et l’analyse de données.
Une brève introduction
L’objectif de ce post est de se familiariser avec différents usages d’Arrow et DuckDB en les manipulant, sur des exemples simples qui peuvent être utile dans un processus de traitement de données. Mais avant de regarder des exemples, qu’est-ce qu’Apache Arrow et DuckDB ?
Apache Arrow est une plateforme qui définit un format de données en mémoire, multi langage et en colonne. Il permet transférer des données sans copie en supprimant le besoin de sérialisation. Arrow est souvent associé au format parquet qui permet de stocker des données tabulaires.
DuckDB est un système de gestion de base de données, en colonne, ne nécessitant aucune installation (à la manière de SQLite). C’est un système OLAP (Online Analytical Processing), contrairement à SQLite qui est un OLTP (Online Transactional Processing). DuckDB possède son format de stockage (.duckdb), mais son moteur peut également être utilisé directement sur des objets Arrow ou des fichiers .parquet.
Ils permettent donc tous les deux de traiter des données en colonnes, par opposition au format .csv ou aux bases OLTP qui traitent les données en ligne, et sont multiplateforme, multi langages. Ils permettent également de retourner le résultat de requêtes sans avoir à charger toutes les données en mémoire.
Dans un premier temps, je vais montrer différents cas d’utilisation, comment lire, écrire, requêter avec Arrow et DuckDB sur un jeu de données simple (parlmerpenguins). Ce jeu de données est petit, il tient donc en mémoire, mais le but ici est de se familiariser avec Arrow et DuckDB, sur des données accessibles à tous, sans se préoccuper de télécharger des grosses quantités de données.
Dans un deuxième temps, je passerais sur un jeu de données plus conséquent (59 Millions d’observations) pour faire un benchmark des différents formats (.csv, .parquet, .duckdb) et des temps de requête en fonction des moteurs et des formats.
Avant de commencer, chargeons-les packages nécessaires pour ce post :
library(arrow)
library(lobstr)
library(tictoc)
library(fs)
library(palmerpenguins)
library(duckdb)
library(dplyr)
Utilisation d’Arrow
Premier contact
En chargeant la librairie palmerpenguins, le dataframe penguins est disponible. Il est composé de 8 colonnes et 344 observations.
glimpse(penguins)Rows: 344
Columns: 8
$ species <fct> Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adelie,…
$ island <fct> Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, Torgersen, T…
$ bill_length_mm <dbl> 39.1, 39.5, 40.3, NA, 36.7, 39.3, 38.9, 39.2, 34.1, 42.0, 37.8, 37.8, 41.1, 38.6, 34.6, 36.6, 38.7, 42.5, 34.4,…
$ bill_depth_mm <dbl> 18.7, 17.4, 18.0, NA, 19.3, 20.6, 17.8, 19.6, 18.1, 20.2, 17.1, 17.3, 17.6, 21.2, 21.1, 17.8, 19.0, 20.7, 18.4,…
$ flipper_length_mm <int> 181, 186, 195, NA, 193, 190, 181, 195, 193, 190, 186, 180, 182, 191, 198, 185, 195, 197, 184, 194, 174, 180, 18…
$ body_mass_g <int> 3750, 3800, 3250, NA, 3450, 3650, 3625, 4675, 3475, 4250, 3300, 3700, 3200, 3800, 4400, 3700, 3450, 4500, 3325,…
$ sex <fct> male, female, female, NA, female, male, female, male, NA, NA, NA, NA, female, male, male, female, female, male,…
$ year <int> 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 200…On peut faire une requête en utilisant dplyr, et on va obtenir immédiatement le résultat :
penguins |>
group_by(species, sex) |>
summarise(avg_body_mass = mean(body_mass_g, na.rm = TRUE)) |>
ungroup()# A tibble: 8 × 3
species sex avg_body_mass
<fct> <fct> <dbl>
1 Adelie female 3369.
2 Adelie male 4043.
3 Adelie NA 3540
4 Chinstrap female 3527.
5 Chinstrap male 3939.
6 Gentoo female 4680.
7 Gentoo male 5485.
8 Gentoo NA 4588.Comment faire la même requête en utilisant Arrow ?
Avant de voir comment utiliser Arrow avec des données volumineuses stockées sur disque, on va reproduire la requête précédente avec Arrow. Pour cela, on créer un objet arrow à partir de ce dataframe avec la fonction arrow::arrow_table() :
penguins_arrow <- arrow_table(penguins)
penguins_arrowTable
344 rows x 8 columns
$species <dictionary<values=string, indices=int8>>
$island <dictionary<values=string, indices=int8>>
$bill_length_mm <double>
$bill_depth_mm <double>
$flipper_length_mm <int32>
$body_mass_g <int32>
$sex <dictionary<values=string, indices=int8>>
$year <int32>L’objet créé contient la définition de la table, mais ne contient aucune donnée à ce stade.
Comme pour un dataframe, on peut utiliser dplyr avec un objet Arrow. On reproduit donc la requête précédente :
penguins_arrow |>
group_by(species, sex) |>
summarise(avg_body_mass = mean(body_mass_g, na.rm = TRUE)) |>
ungroup()Table (query)
species: dictionary<values=string, indices=int8>
sex: dictionary<values=string, indices=int8>
avg_body_mass: double
* Grouped by species
See $.data for the source Arrow objectLà encore, aucune donnée n’est renvoyée. Nous avons juste défini la requête pour être évaluée, mais il faut demander explicitement l’exécution de la requête. Il y a deux fonctions pour cela compute() et collect().
compute(): exécute la requête et les données sont stockées dans l’objet Arrow.collect(): exécute la requête et retourne les données dans R sous forme de tibble.
penguins_arrow |>
group_by(species, sex) |>
summarise(avg_body_mass = mean(body_mass_g, na.rm = TRUE)) |>
collect()# A tibble: 8 × 3
# Groups: species [3]
species sex avg_body_mass
<fct> <fct> <dbl>
1 Adelie male 4043.
2 Adelie female 3369.
3 Adelie NA 3540
4 Gentoo female 4680.
5 Gentoo male 5485.
6 Chinstrap female 3527.
7 Chinstrap male 3939.
8 Gentoo NA 4588.Nous avons pu utiliser la syntaxe habituelle dans R avec dplyr pour requêter un objet Arrow comme on le fait pour un dataframe dans R.
Fichier Parquet
On a vu un exemple simple pour créer un objet Arrow à partir d’un tibble, et une requête en utilisant dplyr pour se familiariser avec la syntaxe. Cependant, le tibble était chargé en mémoire, ce qui nous intéresse c’est de requêter sans avoir à charger les données en mémoire mais à partir de données sur disque. On va donc s’intéresser au format de fichier parquet.
Parquet est un format pour stocker des données en colonne, avec compression et encodage des données, et qui a des performances améliorées pour accéder aux données.
On peut sauvegarder un dataframe ou une table Arrow au format parquet avec write_parquet()
write_parquet(penguins_arrow, here::here("data", "penguins.parquet"))La fonction write_dataset() apporte un format de stockage plus efficace et en particulier peut partitionner les données en fonction d’une variable, en utilisant le style Hive.
Par exemple, notre dataframe contient 3 espèces de pingouins. On peut décider de partitionner en autant de fichiers qu’il y a d’espèce. Ainsi, 3 fichiers seront créés avec une arborescence par espèce, et les fichiers n’auront pas besoin de contenir la variable espèce avec la valeur qui se répète pour chaque observation, ce qui économise de la place. C’est également plus rapide lors de l’analyse d’un sous-ensemble pour une seule espèce, puisqu’Arrow n’accédera qu’à un seul fichier.
write_dataset(penguins,
here::here("data", "penguins_species"),
format = "parquet",
partitioning = c("species"))La structure sur le disque est la suivante :
dir_tree(here::here("data", "penguins_species"))D:/env/data-pipeline/data/penguins_species
├── species=Adelie
│ └── part-0.parquet
├── species=Chinstrap
│ └── part-0.parquet
└── species=Gentoo
└── part-0.parquetMaintenant que nous disposons des données au format parquet, on va refaire le workflow précédent, en pointant sur le fichier sur disque plutôt et sans charger les données en mémoire dans R.
On ouvre un objet arrow dataset qui pointe sur le dataset multi-fichiers qu’on vient créer.
penguins_arrow <- open_dataset(here::here("data", "penguins_species"))
penguins_arrowFileSystemDataset with 3 Parquet files
island: dictionary<values=string, indices=int32>
bill_length_mm: double
bill_depth_mm: double
flipper_length_mm: int32
body_mass_g: int32
sex: dictionary<values=string, indices=int32>
year: int32
species: stringComme prévu, penguins_arrow ne contient aucune donnée, mais est un objet qui va nous permettre de faire une requête.
penguins_arrow |>
group_by(species, sex) |>
summarise(avg_body_mass = mean(body_mass_g, na.rm = TRUE)) |>
collect()# A tibble: 8 × 3
# Groups: species [3]
species sex avg_body_mass
<chr> <fct> <dbl>
1 Adelie male 4043.
2 Adelie female 3369.
3 Adelie NA 3540
4 Chinstrap female 3527.
5 Chinstrap male 3939.
6 Gentoo female 4680.
7 Gentoo male 5485.
8 Gentoo NA 4588.On obtient un tibble contenant uniquement le résultat, les données contenues dans les fichiers parquet n’ont pas été chargés en mémoire dans R. Bien sûr ce n’est pas perceptible sur un dataframe si petit, mais on verra plus loin l’impact sur des données plus volumineuses.
Utilisation de DuckDB
Avec Arrow & dplyr
On peut utiliser le moteur de DuckDB pour requêter un objet Arrow avec dplyr. Rien de plus simple, il suffit d’utiliser la fonction to_duckdb():
penguins_arrow |>
to_duckdb() |>
group_by(species, sex) |>
summarise(avg_body_mass = mean(body_mass_g, na.rm = TRUE))# Source: lazy query [?? x 3]
# Database: DuckDB 0.5.1 [christophe@Windows 10 x64:R 4.2.0/:memory:]
# Groups: species
species sex avg_body_mass
<chr> <chr> <dbl>
1 Adelie male 4043.
2 Adelie female 3369.
3 Adelie NA 3540
4 Chinstrap female 3527.
5 Chinstrap male 3939.
6 Gentoo female 4680.
7 Gentoo male 5485.
8 Gentoo NA 4588.On constate que le résultat est retourné en mentionnant “# Database: DuckDB 0.5.1”, c’est bien le moteur de DuckDB qui a été utilisé.
Un des intérêts de passer par le moteur de DuckDB et dplyr peut être d’utiliser une fonctionnalité implémentée par DuckDB mais pas encore par Arrow. On peut faire la démarche inverse, et revenir au moteur d’Arrow avec to_arrow().
L’interopérabilité entre Arrow, DuckDB et dplyr est très simple à utiliser et apporte beaucoup de flexibilité pour le traitement des données.
Avec Arrow & SQL
Grace à DuckDB, on peut utiliser SQL directement sur un objet Arrow pour effectuer la requête.
On créer la connexion avec la base de données, puis on enregistre l’objet arrow comme table virtuelle DuckDB, en lui donnant un nom qui sera utilisé dans la requête SQL, enfin on exécute la requête.
con <- dbConnect(duckdb::duckdb())
arrow::to_duckdb(penguins_arrow, table_name = "penguins", con = con)
dbGetQuery(con, "SELECT species, sex, AVG(body_mass_g) FROM penguins GROUP BY species, sex") species sex avg(body_mass_g)
1 Adelie male 4043.493
2 Adelie female 3368.836
3 Adelie <NA> 3540.000
4 Gentoo female 4679.741
5 Gentoo male 5484.836
6 Gentoo <NA> 4587.500
7 Chinstrap female 3527.206
8 Chinstrap male 3938.971Une fois toutes les manipulations effectuées, ne pas oublier de fermer la connexion :
duckdb_unregister(con, "penguins")
dbDisconnect(con)
Avec Parquet et SQL
Il est également possible d’accéder à un fichier Parquet directement dans la requête SQL, sans passer par la création d’un objet Arrow, en utilisant read_parquet():
con <- dbConnect(duckdb::duckdb())
dbGetQuery(con, "SELECT species, sex, AVG(body_mass_g) FROM read_parquet('penguins.parquet') GROUP BY species, sex")
dbDisconnect(con) species sex avg(body_mass_g)
1 Adelie male 4043.493
2 Adelie female 3368.836
3 Adelie <NA> 3540.000
4 Gentoo female 4679.741
5 Gentoo male 5484.836
6 Gentoo <NA> 4587.500
7 Chinstrap female 3527.206
8 Chinstrap male 3938.971read_parquet() peut lire un fichier Parquet ou une liste de fichiers, cependant, DuckDB ne peut pas encore ouvrir des fichiers multi-niveaux créés en partitionnant. Il faut donc pour cela passer par Arrow et ouvrir le dataset, comme vu précédemment.
De la même façon, DuckDB peut lire des fichiers CSV directement, ou même créer une table à partir d’un fichier CSV, ce qui est très intéressant pour importer des données dans une table DuckDB.
En effet, DuckDB est une base de données et a donc également son propre format de stockage.
Format .duckdb
Le stockage de DuckDB va plus loin que le format Parquet. En effet, il permet de stocker une base de données avec de multiples tables, views, supporte les opérations ACID, l’altération des tables, ajouts de colonnes sans avoir à réécrire entièrement le fichier.
Pour créer la base depuis R, on utilise la fonction duckdb(), on peut créer la base de données directement en créant la connection avec dbConnect() comme ci-dessous.
con <- dbConnect(duckdb::duckdb(), dbdir="penguins.duckdb", read_only=FALSE)On peut alors créer la table penguins avec les données à partir d’un dataframe avec la syntaxe suivante :
duckdb::dbWriteTable(con, "penguins", penguins)On peut également créer la table avec une requête SQL en important les données directement à partir d’un fichier, par exemple Parquet ou csv :
dbSendQuery(con, "CREATE TABLE penguins_parquet AS SELECT * FROM read_parquet('penguins.parquet');")Ou encore à partir d’un objet Arrow, en enregistrant l’objet Arrow comme table virtuelle, et en créant la table DuckDb à partir de cette table :
arrow::to_duckdb(penguins_arrow, table_name = "penguins_arrow", con = con)
dbSendQuery(con, "CREATE TABLE penguins_arrow AS SELECT * FROM penguins_arrow")On peut lister les tables contenues dans la base avec la commande suivante :
dbListTables(con)[1] "penguins" "penguins_arrow" "penguins_parquet"Les possibilités sont très intéressantes dès lors qu’on parle d’utiliser de très grosses quantités de données qui ne tiennent pas en mémoire. On peut ainsi alimenter la base de données à partir de fichiers parquet volumineux en passant par Arrow, sans avoir à charger les données en mémoire dans R.
L’intérêt d’utiliser une base duckDB, outre les raisons précitées, est que les requêtes sont beaucoup plus rapides qu’à partir d’un fichier Parquet. Bien entendu cela a un coût en termes de stockage. En effet, le format Parquet est plus compressé, et prend moins de place sur disque par rapport à une base DuckDB. Il y a donc un choix à faire, si on privilégie la vitesse, la compression, et si on a besoin des fonctionnalités d’une base de données.
Comparatif des format et moteurs
Préparation des données
On s’est concentré sur le fonctionnement d’Arrow et DuckDB avec R sur un jeu de donnée simples. A présent, regardons les performances sur deux critères : la taille sur disque et la vitesse.
Pour cela je vais utiliser un jeu de donnée plus volumineux, préparé à partir de fichiers présents sur Kaggle.
Si vous voulez reproduire, les fichiers sont téléchargeables sur Kaggle, et voici le code pour générer le dataframe.
sales <- read_csv(here::here("data", "M5", "sales_train_evaluation.csv"))
calendar <- read_csv(here::here("data", "M5", "calendar.csv"))
sell_prices <- read_csv(here::here("data", "M5", "sell_prices.csv"))
sales <- sales |>
pivot_longer(cols=contains("d_"), names_to = "d", values_to = "qty")
sales <- sales |>
left_join(calendar, by = "d") |>
left_join(sell_prices, by = c("wm_yr_wk", "store_id", "item_id")) |>
replace_na(list(sell_price = 0))A noter que dans une utilisation normale, il serait plus judicieux de stocker chaque fichier dans une table différente et de faire la jointure avec Arrow ou DuckDB, mais l’objectif ici est de créer un dataframe volumineux.
De plus les données brutes prennent environ 8 GB ce qui techniquement tient toujours en mémoire sur des PC actuels, mais me permet de stocker les différents formats sur mon disque, tout en appréciant les différences de performances.
Taille des différents formats de fichier
On va créer 4 formats différents, csv, Parquet créé à partir d’un csv (pour illustrer un point important), Parquet créé à partir d’un dataframe et DuckDB.
Sauvegarde au format csv :
write_csv(sales, here::here("data", "sales.csv"))Sauvegarde au format Parquet à partir d’un fichier csv en utilisant Arrow :
sales_csv_arrow <- open_dataset(here::here("storage", "sales.csv"), format = "csv")
write_dataset(sales_csv_arrow,
here::here("storage", "sales_csv_parquet"),
format = "parquet",
partitioning = c("year", "month"))Sauvegarde au format Parquet avec partionnement à partir du dataframe :
write_dataset(sales,
here::here("storage", "sales_parquet"),
format = "parquet",
partitioning = c("year", "month"))Création de la base DuckDB à partir du format Parquet partionné :
sales_arrow <- open_dataset(here::here("storage", "sales_parquet"))
con <- dbConnect(duckdb::duckdb(), dbdir=here::here("storage", "sales_duckdb", "sales.duckdb"), read_only=FALSE)
arrow::to_duckdb(sales_arrow, table_name = "sales", con = con)
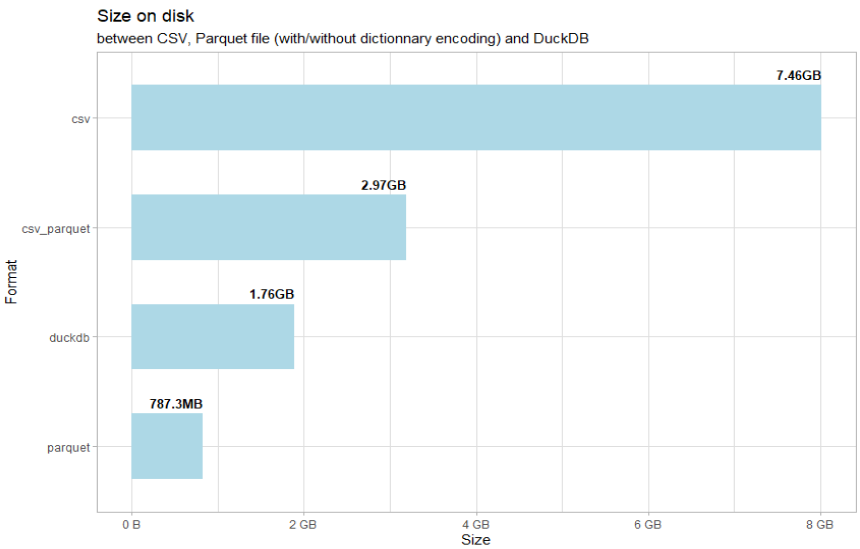
dbSendQuery(con, "CREATE TABLE daily_sales AS SELECT * FROM sales")Comparons les tailles sur disque :
disk_size <- dir_info(here::here("storage"), recurse = TRUE) |>
mutate(relative_path = str_remove(path, here::here("storage")),
format = str_match(relative_path, "/(.+?)/")[,2],
format = str_remove(format, "sales_")) |>
arrange(format) |>
group_by(format) |>
summarise(total = sum(size)) |>
ungroup() |>
drop_na() |>
mutate(format = forcats::fct_reorder(format, total))
disk_size |>
ggplot(aes(format, total)) +
geom_col(fill = "lightblue", width=0.6)+
geom_text(aes(label = glue::glue("{total}B")), hjust = 1, nudge_x = 0.4, size = 3.5, fontface = "bold") +
coord_flip()+
scale_y_continuous(labels = scales::label_bytes(units = "auto_si", accuracy = 1))+
labs(title = "Size on disk",
subtitle ="between CSV, Parquet file (with/without dictionnary encoding) and DuckDB",
x = "Format",
y = "Size")+
theme_light()On voit que le format csv prend près de 8 GB. Le format DuckDB est près de 4 fois plus petit, et Parquet près de 10 fois plus léger.
Pourquoi le format Parquet créé directement à partir du CSV est-il plus volumineux ?
Parquet utilise par défaut un encodage avec un dictionnaire pour représenter les chaines de caractères. Il créé un dictionnaire pour conserver l’association clé (format integer) et valeur (format string), et ne stock ainsi les données que sous forme d’integer beaucoup moins volumineux qu’une chaine de caractère.
Prenons l’exemple de la variable cat_id. Elle contient uniquement 3 valeurs uniques (“HOBBIES”, “HOUSEHOLD”, “FOODS”) qui se répètent 59 millions de fois. Plutôt que de stocker 59 millions de chaines de caractère, Parquet utilise un dictionnaire pour associer chaque chaine à un integer (0=“HOBBIES”, 1=“HOUSEHOLD”, 2=“FOODS”), et ne stocke que la valeur de l’integer.
Dans le cas de la création du fichier Parquet à partir du dataframe en mémoire, Arrow est capable de trouver les valeurs uniques pour créer le dictionnaire, et utilise donc l’encodage avec dictionnaire.
Dans le cas d’une création à partir du fichier csv, il n’est pas possible de connaître le nombre de valeurs uniques avant d’avoir traité la totalité du fichier csv, il utilise donc le format le plus simple qui est de stocker les chaines de caractères sous forme de chaines de caractère.
Ceci est transparent pour l’utilisateur, si on regarde le schéma, le type de la variable cat_id est toujours “string” :
sales_arrow$schemaSchema
id: string
item_id: string
dept_id: string
cat_id: string
store_id: string
state_id: string
d: string
qty: double
date: date32[day]
wm_yr_wk: double
weekday: string
wday: double
event_name_1: string
event_type_1: string
event_name_2: string
event_type_2: string
snap_CA: double
snap_TX: double
snap_WI: double
sell_price: double
year: int32
month: int32A noter qu’il existe également un type dictionnaire. Par exemple, si on utilise des factors, ce qui est le cas pour les variables species, island et sex du jeu de données penguins, on voit que les factors sont stockés avec le type de données dictionnaire.
arrow_table(penguins)Table
344 rows x 8 columns
$species <dictionary<values=string, indices=int8>>
$island <dictionary<values=string, indices=int8>>
$bill_length_mm <double>
$bill_depth_mm <double>
$flipper_length_mm <int32>
$body_mass_g <int32>
$sex <dictionary<values=string, indices=int8>>
$year <int32>Je ne développe pas plus ici les aspects de type, schéma etc., mais c’est important d’y être sensible au moins pour deux raisons :
- Utiliser le type le plus approprié : par exemple si un integer ne prend qu’une valeur comprise entre 0 et 255, utiliser int8 plutôt que int32. Les utilisateurs de Python sont déjà sensibles à ce type de conversion, mais c’est moins le cas avec R, avec lequel on utilise juste integer, double sans notion de int8, int16, int32 ou float16, float32, float64.
- Jointure : La jointure entre deux fichiers va échouer si les types de données utilisés pour la jointure sont différents, il faut alors convertir une des deux variables pour qu’elles aient le même type.
Performance des requêtes
On va comparer les performances sur une requête permettant de calculer les revenus mensuels par catégorie et par état, pour l’année 2015. Ça permet d’avoir à la fois un filtre, un group by, un calcul, et un tri.
Pour le fichier CSV, j’ai séparé le temps de lecture des données et de la requête :
tic()
sales <- read_csv(here::here("storage", "sales_csv", "sales.csv"))
toc()
tic()
sales |>
filter(year==2015) |>
group_by(state_id, cat_id, month) |>
summarise(monthly_revenue = sum(qty * sell_price, na.rm = TRUE)) |>
ungroup() |>
arrange(cat_id, month)
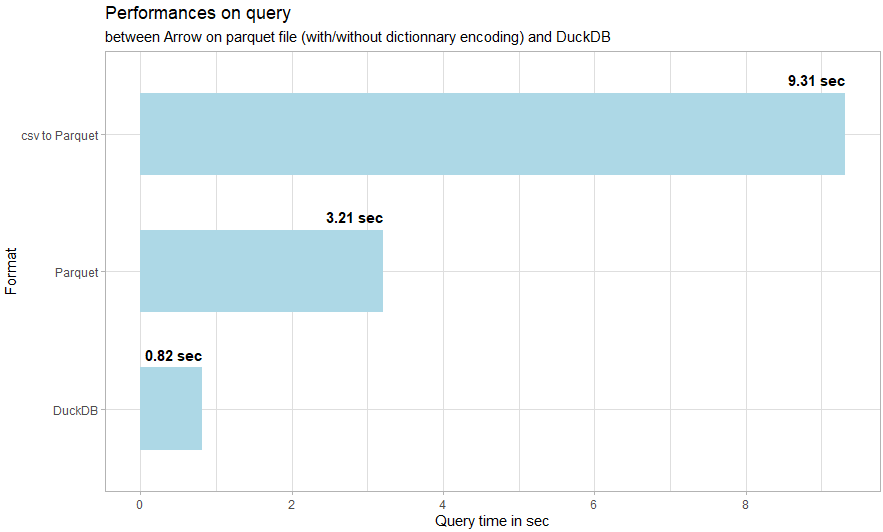
toc()2258.86 sec elapsed
44.07 sec elapsed
Créer cette requête sur un fichier csv prend en tout 2302.93 secondes, soit plus de 38 minutes, dont 37 pour la lecture des données, et 44 pour la requête une fois les données chargées en mémoire.
Pour le parquet sans variable catégorielle :
tic()
sales_arrow <- open_dataset(here::here("storage", "sales_csv_parquet"))
sales_arrow |>
filter(year==2015) |>
group_by(state_id, cat_id, month) |>
summarise(monthly_revenue = sum(qty * sell_price, na.rm = TRUE)) |>
ungroup() |>
arrange(cat_id, month) |>
collect()
toc()9.31 sec elapsed
Pour le fichier parquet avec variables catégorielles :
tic()
sales_arrow <- open_dataset(here::here("storage", "sales_parquet"))
sales_arrow |>
filter(year==2015) |>
group_by(state_id, cat_id, month) |>
summarise(monthly_revenue = sum(qty * sell_price, na.rm = TRUE)) |>
ungroup() |>
arrange(state_id,cat_id, month) |>
collect()
toc()3.21 sec elapsed
Et enfin DuckDB :
tic()
con <- dbConnect(duckdb::duckdb(), dbdir=here::here("storage", "sales_duckdb", "sales.duckdb"), read_only=TRUE)
dbListTables(con)
dbGetQuery(con, "SELECT state_id, cat_id, month, SUM(qty * sell_price) FROM daily_sales WHERE year = 2015 GROUP BY state_id, cat_id, month ORDER BY state_id, cat_id, month")
duckdb::dbDisconnect(con)
toc()0.82 sec elapsed
On voit clairement que le format csv est très loin au niveau des performances, avec 2303 secondes, soit plus de 38 minutes. Je l’exclue donc de la visualisation pour pouvoir plus aisément visualiser les différences entre parquet_csv, parquet et duckdb :
performance <- tibble(format = c("csv", "csv_parquet", "parquet", "duckdb"),
format_text = c("csv", "csv to Parquet", "Parquet", "DuckDB"),
elapse_time = c(2302.93, 9.31, 3.21, 0.82))
performance |>
filter(format != "csv") |>
mutate(format_text = forcats::fct_reorder(format_text, elapse_time)) |>
ggplot(aes(format_text, elapse_time))+
geom_col(fill = "lightblue", width=0.6)+
geom_text(aes(label = glue::glue("{elapse_time} sec")), hjust = 1, nudge_x = 0.4, size = 4, fontface = "bold") +
coord_flip()+
scale_y_continuous(breaks=seq(0,10,2)) +
labs(title = "Performances on query",
subtitle ="between Arrow on parquet file (with/without dictionnary encoding) and DuckDB",
x = "Format",
y = "Query time in sec")+
theme_light()Concernant les performances, parquet est 717 fois plus rapide que la même requête sur un fichier csv, et duckdb est 2808 fois plus rapide.
DuckDB lui est 4 fois plus rapide que la requête sur une fichier parquet. Gardons à l’esprit qu’il y a un compromis à faire puisque le fichier parquet est plus compressé et prend 2.5 fois moins de place que la base au format duckdb.
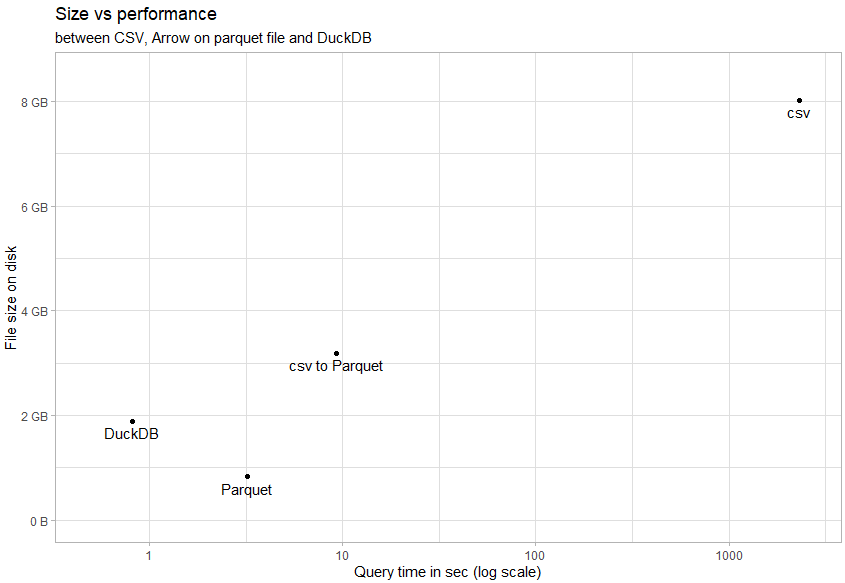
On peut visualiser un résumé avec le compromis taille / performance (échelle logarithmique) :
disk_size |>
left_join(performance) |>
ggplot()+
geom_point(aes(elapse_time, total)) +
geom_text(aes(elapse_time, total, label = format_text), nudge_y = -1e8, vjust = 1) +
scale_x_log10(limits=c(0.5,2500))+
scale_y_continuous(labels = scales::label_bytes(units = "auto_si", accuracy = 1), limits = c(0.5e9,8.5e9)) +
labs(title = "Size vs performance",
subtitle ="between CSV, Arrow on parquet file and DuckDB",
x = "Query time in sec (log scale)",
y = "File size on disk")+
theme_light()
Utilisation de la mémoire
On a pu voir au niveau des performances que pour le cas d’un fichier csv, toutes les données sont chargées en mémoire, alors que pour Arrow et DuckDB, seul le résultat est retourné.
On peut également s’en assurer en regardant la taille des objets :
Fichier csv chargé en mémoire :
str_obj(sales)10.42 GB
Utilisation d’Arrow:
str_obj(sales_arrow)261.59 kB
Lors de l’exécution de la requête sur les données provenant du fichier csv, la mémoire utilisée par R sur mon laptop dépassais 13 GB (taille du dataframe + requête), alors qu’elle est restée inférieure à 1 GB pour la requête avec Arrow.
Conclusion
On a vu comment utiliser Apache Arrow et DuckDB pour des manipulations courantes, passer d’un moteur à l’autre, d’un format à l’autre, utiliser dplyr ou SQL, et enfin voir les bénéfices sur l’espace de stockage, de performance pour les requêtes, sans charger les données en mémoire dans R.
On a donc tous les éléments pour définir l’architecture la plus adaptée pour collecter et analyser des données volumineuses.
En récapitulant les avantages :
- Interopérabilité entre Arrow, DuckDB et dplyr
- Analyse de données qui ne tiennent pas en mémoire
- Formats compressés plus légers en place que le traditionnel CSV
- Performance en temps de requête
- Multiplateforme et multilangage, vous pouvez utiliser le dataset ou la base de données DuckDB créés avec R sous Windows sans problème avec Python ou Julia sous Linux
J’ai utilisé conjointement Apache Arrow et DuckDB pour manipuler des jeu de données de grande taille. Cependant, il faut noter que DuckDB est un système de gestion de base de donnée OLAP qui apporte plus de fonctionnalités qu’Apache Arrow pour batir une solution analytique.
Note : Je me suis focalisé uniquement sur le format Parquet pour ne pas surcharger le post, mais Apache Arrow utilise le format Feather, plus léger, mais moins compressé.